[ad_1]
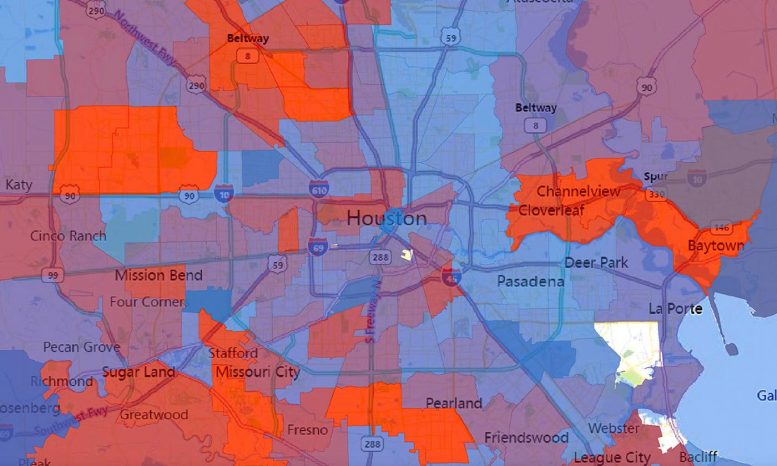
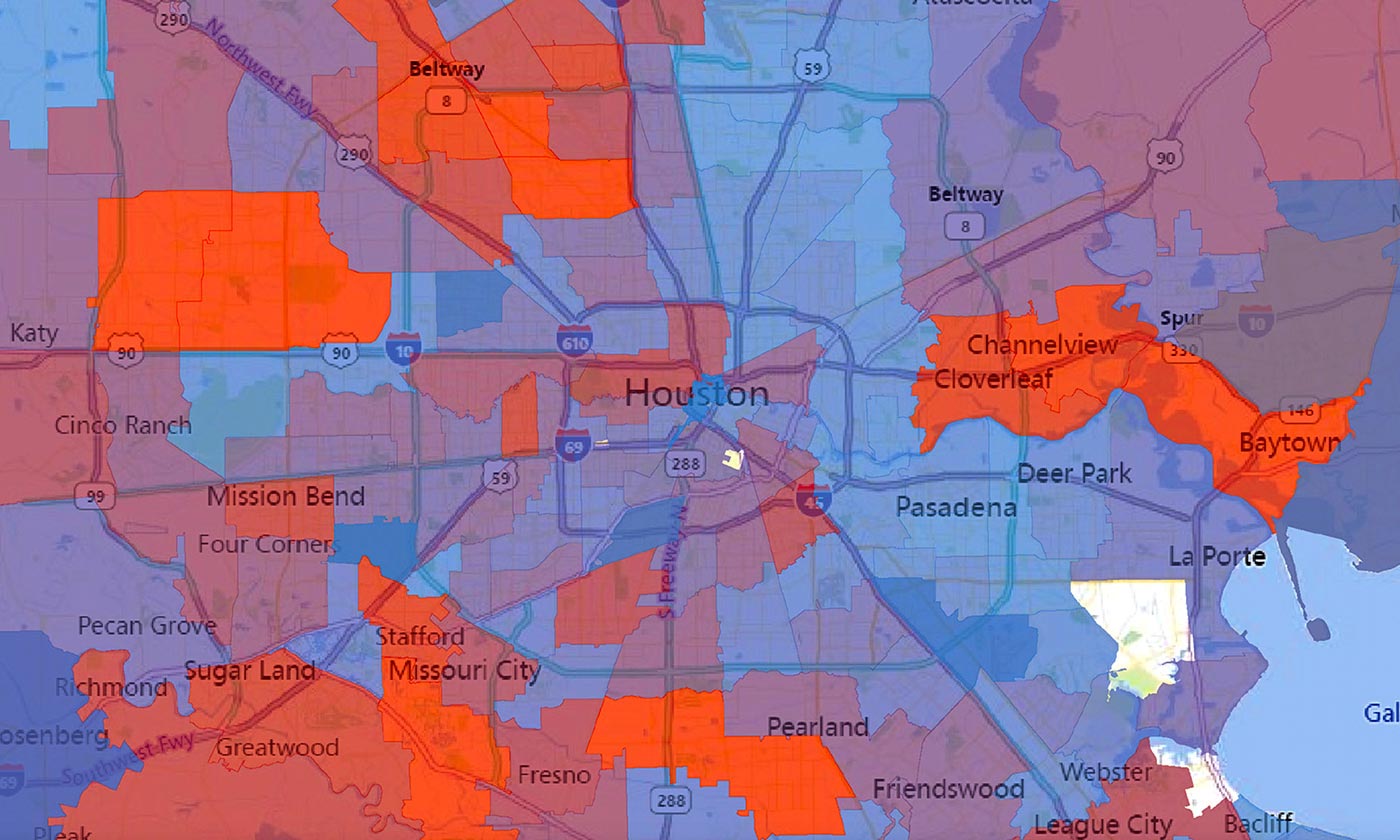
The number of viral strains present in each zip code in Houston during the second wave of COVID-19 cases in the summer of 2020. The number of strains is represented by a spectrum of colors ranging from blue (0 strain) to red ( 50 strains). Credit: Houston Methodist / University of Texas at Austin. Credit: Houston Methodist / University of Texas at Austin
A study of more than 5,000 COVID-19[feminine les patients de Houston constatent que le virus qui cause la maladie accumule des mutations génétiques, dont l’une peut l’avoir rendue plus contagieuse. Selon l’article publié dans la revue à comité de lecture mBIO, cette mutation, appelée D614G, est située dans la protéine de pointe qui ouvre nos cellules à l’entrée virale. Il s’agit de la plus grande étude évaluée par des pairs SRAS-CoV-2 séquences génomiques dans une région métropolitaine des États-Unis à ce jour.
L’article montre que «le virus mute en raison d’une combinaison de dérive neutre – ce qui signifie simplement des changements génétiques aléatoires qui n’aident pas ou ne nuisent pas au virus – et la pression de notre système immunitaire», a déclaré Ilya Finkelstein, professeur agrégé de biosciences moléculaires L’Université du Texas à Austin et co-auteur de l’étude. L’étude a été réalisée par des scientifiques de l’hôpital méthodiste de Houston, de l’UT Austin et ailleurs.
Au cours de la vague initiale de la pandémie, 71% des nouveaux coronavirus identifiés chez les patients de Houston avaient cette mutation. Lorsque la deuxième vague de l’épidémie a frappé Houston au cours de l’été, cette variante avait bondi à 99,9% de prévalence. Cela reflète une tendance observée dans le monde entier. Une étude publiée en juillet sur la base de plus de 28 000 séquences génomiques a révélé que les variants porteurs de la mutation D614G sont devenus la forme dominante mondiale de SRAS-CoV-2 en un mois environ. Le SARS-CoV-2 est le coronavirus qui cause le COVID-19.
Alors pourquoi les souches contenant cette mutation ont-elles surpassé celles qui ne l’avaient pas?
Peut-être sont-ils plus contagieux. Une étude portant sur plus de 25 000 séquences génomiques au Royaume-Uni a révélé que les virus porteurs de la mutation avaient tendance à se transmettre légèrement plus rapidement que ceux qui n’en avaient pas et provoquaient de plus grandes grappes d’infections. La sélection naturelle favoriserait les souches du virus qui se transmettent plus facilement. Mais tous les scientifiques ne sont pas convaincus. Certains ont suggéré une autre explication, appelée «effets du fondateur». Dans ce scénario, la mutation D614G aurait pu être plus courante dans les premiers virus à arriver en Europe et en Amérique du Nord, leur donnant essentiellement une longueur d’avance sur d’autres souches.
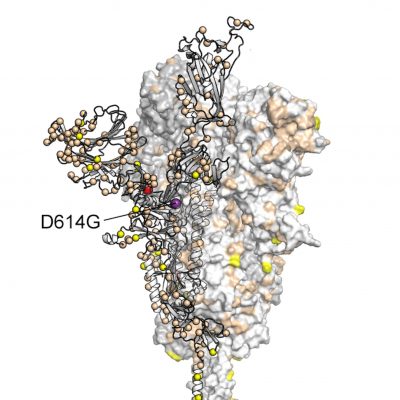
La plupart des souches de coronavirus circulant à Houston à l’été 2020 avaient la mutation D614G dans la protéine de pointe. Crédit: Méthodiste de Houston / Université du Texas à Austin
La protéine de pointe continue également d’accumuler des mutations supplémentaires de signification inconnue. L’équipe Houston Methodist-UT Austin a également montré dans des expériences de laboratoire qu’au moins une de ces mutations permet à Spike d’échapper à un anticorps neutralisant que les humains produisent naturellement pour lutter contre les infections par le SRAS-CoV-2. Cela peut permettre à cette variante du virus de passer plus facilement au-delà de notre système immunitaire. Bien qu’il ne soit pas encore clair si cela se traduit par une transmission plus facile entre les individus.
La bonne nouvelle est que cette mutation est rare et ne semble pas aggraver la maladie chez les patients infectés. Selon Finkelstein, le groupe n’a pas vu de virus qui ont appris à échapper aux vaccins de première génération et aux formulations d’anticorps thérapeutiques.
«Le virus continue de muter à mesure qu’il traverse le monde», a déclaré Finkelstein. «Les efforts de surveillance en temps réel comme notre étude garantiront que les vaccins et les thérapies mondiales ont toujours une longueur d’avance.»
Les scientifiques ont noté un total de 285 mutations sur des milliers d’infections, bien que la plupart ne semblent pas avoir d’effet significatif sur la gravité de la maladie. Des études en cours se poursuivent pour surveiller la troisième vague de patients COVID-19 et pour caractériser la façon dont le virus s’adapte aux anticorps neutralisants produits par notre système immunitaire. Chaque nouvelle infection est un jet de dés, une chance supplémentaire de développer des mutations plus dangereuses.
«Nous avons donné beaucoup de chances à ce virus», a déclaré l’auteur principal James Musser de Houston Methodist au Washington Post.. «Il y a une population énorme en ce moment.»
Plusieurs autres auteurs d’UT Austin ont contribué aux travaux: le chercheur invité Jimmy Gollihar, le professeur agrégé de biosciences moléculaires Jason S. McLellan et les étudiants diplômés Chia-Wei Chou, Kamyab Javanmardi et Hung-Che Kuo.
L’équipe d’UT Austin a testé différentes variantes génétiques de la protéine de pointe du virus, la partie qui lui permet d’infecter les cellules hôtes, de mesurer la stabilité de la protéine et de voir dans quelle mesure elle se lie à un récepteur sur les cellules hôtes et aux anticorps neutralisants. Plus tôt dans l’année, McLellan et son équipe de l’UT Austin, en collaboration avec des chercheurs des National Institutes of Health, ont développé la première carte 3D de la protéine de pointe de coronavirus pour une innovation qui intègre désormais plusieurs conceptions de vaccins de premier plan.
Les chercheurs ont découvert que le SRAS-CoV-2 a été introduit dans la région de Houston à plusieurs reprises, indépendamment, de diverses régions géographiques, avec des souches virales d’Europe, d’Asie, d’Amérique du Sud et d’ailleurs aux États-Unis. Il y a eu une diffusion communautaire généralisée peu de temps après que les cas de COVID-19 ont été signalés à Houston.
Référence: «Architecture moléculaire de la diffusion précoce et de la deuxième vague massive du virus SRAS-CoV-2 dans une région métropolitaine majeure» par S. Wesley Long, Randall J. Olsen, Paul A. Christensen, David W. Bernard, James J. Davis, Maulik Shukla, Marcus Nguyen, Matthew Ojeda Saavedra, Prasanti Yerramilli, Layne Pruitt, Sishir Subedi, Hung-Che Kuo, Heather Hendrickson, Ghazaleh Eskandari, Hoang AT Nguyen, J. Hunter Long, Muthiah Kumaraswami, Jule, Goike, Daniel Boutz Jimmy Gollihar, Jason S. McLellan, Chia-Wei Chou, Kamyab Javanmardi, Ilya J.Finkelstein et James M. Musser, 30 octobre 2020, mBio.
DOI: 10.1128 / mBio.02707-20
Cette étude a été soutenue par la Fondation Fondren, le Houston Methodist Hospital and Research Institute, les National Institutes of Health, le National Institute of Allergy and Infectious Diseases, la Welch Foundation, la National Science Foundation et la Defense Advanced Research Projects Agency. Ilya Finkelstein est une chercheuse du CPRIT en recherche sur le cancer, financée par le Cancer Prevention and Research Institute of Texas.
Une version antérieure de l’article a été publiée en septembre sur le serveur de pré-impression medRxiv.
[ad_2]
Source link